BioBrick規格について
実験操作や概念の説明、一般編です。
前回の記事で、目的遺伝子の配列を含んだプラスミドを大腸菌に入れることで大腸菌に目的の機能を持たせることができるという話をしました。今回はこのプラスミドについてもう少し詳しく見ていきます。
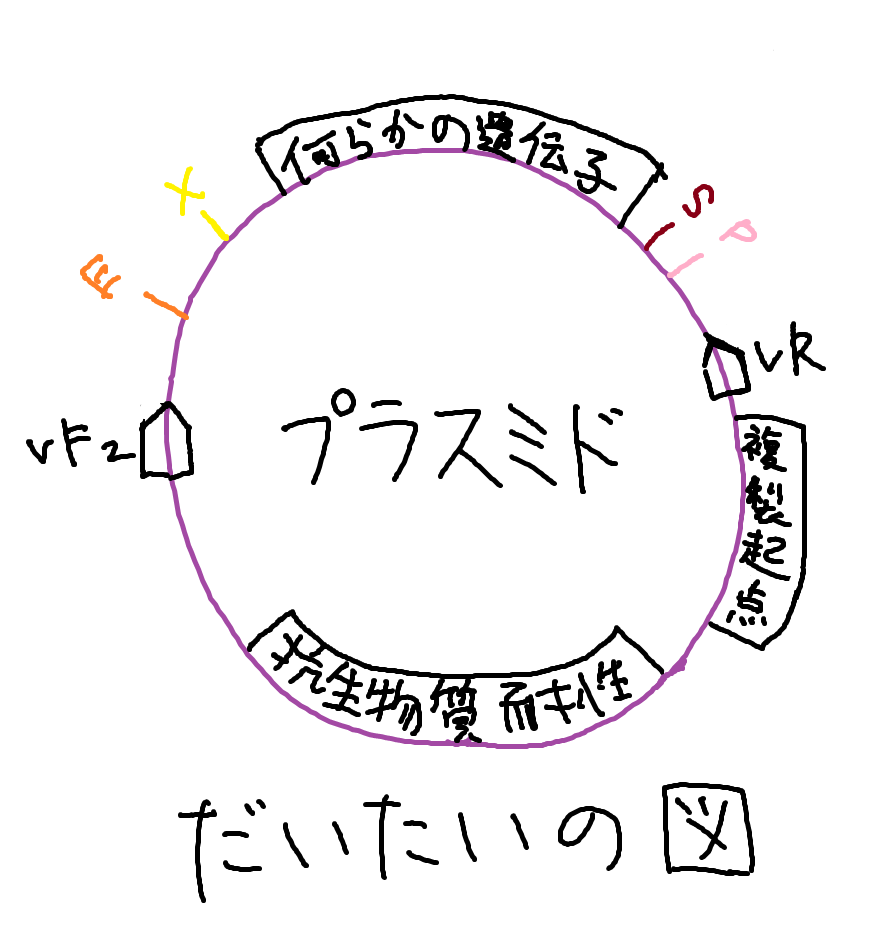
iGEMで遺伝子を導入するために使うプラスミドはBioBrick standardと呼ばれる規格に沿っています。
この図中のそれぞれの要素とその機能などについて触れていきます。
抗生物質耐性遺伝子
この部分には大腸菌に抗生物質に対する耐性を持たせるような遺伝子が来ることになっています。対象となる抗生物質の種類は、クロラムフェニコール、アンピシリン、カナマイシンなど様々で、耐性を持つのはどれか一種類の抗生物質に対してです。
そもそも抗生物質とは細菌の成長を止めたり細菌を殺したりするような化学物質のことで、抗生物質に耐性を持たない大腸菌はその抗生物質の存在下では(原則)生息できません。大腸菌にプラスミドを入れるときにはこのことを利用します。
大腸菌にプラスミドを入れようとしても、培養液にいる大量の大腸菌すべてに100%の確率でプラスミドを入れられるわけではありません。詳しい手法は別の記事で説明しますが、むしろほとんどの大腸菌にはプラスミドが入りません。必要なのはプラスミド入りの大腸菌であるため、何らかの方法で大量のハズレの大腸菌の中からアタリの大腸菌を選び出さなければいけません。このときに抗生物質とそれへの耐性遺伝子が役に立ちます。
ハズレとアタリが混ざっている大腸菌を対応する抗生物質を含んだ培地で培養します。そうすることで、耐性遺伝子を持つ大腸菌、つまりプラスミドが入っている大腸菌だけが生き残り、残りの大腸菌は死にます。培地上で生き残っている大腸菌はプラスミドが入ったアタリの大腸菌ということになり、アタリだけを無事選別できたということになります。
複製起点
大腸菌に入ったプラスミドはそのままその大腸菌に残り続けます。大腸菌は分裂して増えていくわけですが、プラスミドはそのどちらにも引き継がれます。大腸菌はどんどん増えていくのでプラスミドも増える必要がありことがわかります。実際にゲノムDNAが分裂前に複製されて分裂後の二つの細胞にそれぞれ分配されるように、プラスミドも分裂後の2つの細胞に分配されます(プラスミドが複製されるタイミングについては詳しく知りません。そのうち調べておくと思います)。
この分裂が始まる箇所を複製起点といいます。複製起点はbiobrick規格かどうかに関わらず各プラスミドに1つずつ(たぶん)存在し、複製起点の種類によって細胞内でのそのプラスミドの個数(コピー数)が変わります。1細胞あたりコピー数が10になるような複製起点もあれば、1細胞あたりコピー数が1000になるような複製起点も存在します。

図再び
VF2とVR
これはPCRに関わってくるので詳しくは別の記事で説明しますが、biobrickの規格にこれらが含まれていることはどのプラスミドでも同じようにPCRにかけられるというメリットをもたらします。
E, X, S, P
これがbiobrickの規格の中でもっともbiobrickをbiobrickたらしめているものです。ここからの説明は長くなるのでセクションにわけていきます。
制限酵素という酵素が存在します。これは2本鎖DNA(他のものでもいいかもしれない)の特定の塩基配列を認識し特定の箇所でDNAを切断する酵素のことです。切れたDNAをくっつける酵素(リガーゼ)とこの制限酵素をうまいこと使うことで、あるプラスミドの一部を別のプラスミドの一部に挿入することができます。このことが重要です。
各制限酵素認識サイトの説明
図中のE, X, S, PというのはそれぞれEcoR1, Xba1, Spe1, Pst1という名前の制限酵素が認識する塩基配列のことです。このように制限酵素が認識する部位のことを制限酵素認識サイトといいます(他にもいろいろな呼び方がありそう)。
それぞれの認識サイトの具体的な塩基配列を切断箇所は次の図の通りです。

5’と3'いうのはDNAの向きのことです。例えばEcoR1の場合5'-GAATTC-3'は認識するけれど5'-CTTAAG-3'は認識しないといったように、逆向きの配列は認識しません。
これら4つの酵素はどれも特定の6塩基を認識して赤いラインで切断します(他の制限酵素の認識サイトには6塩基ではないものもあるし切断の仕方が全然違うものもあります)。
これら4つを見ると気づくのですが、5'-GAATTC-3'の相補的な配列も5'-GAATTC-3'となっていて、他の3つの制限酵素と同様に認識サイトの相補鎖が認識サイトになっています。このことにより、制限酵素認識サイトがある塩基配列中に存在するかどうかを確かめるときにDNA2本鎖の両方からGAATTCを探す必要がなく、片方の鎖だけを見ればいいことになります、これはいろいろ便利です。(制限酵素認識サイトはこのようになっているものが多いのでたぶん切断する配列を一つにすることが有利になるような淘汰圧が自然界で働いていたんだと思います。)
プラスミドへの遺伝子の挿入方法
制限酵素を使うとプラスミドに遺伝子を挿入することができると言いました。実際にどのような原理でそれができるかを見ていきます。
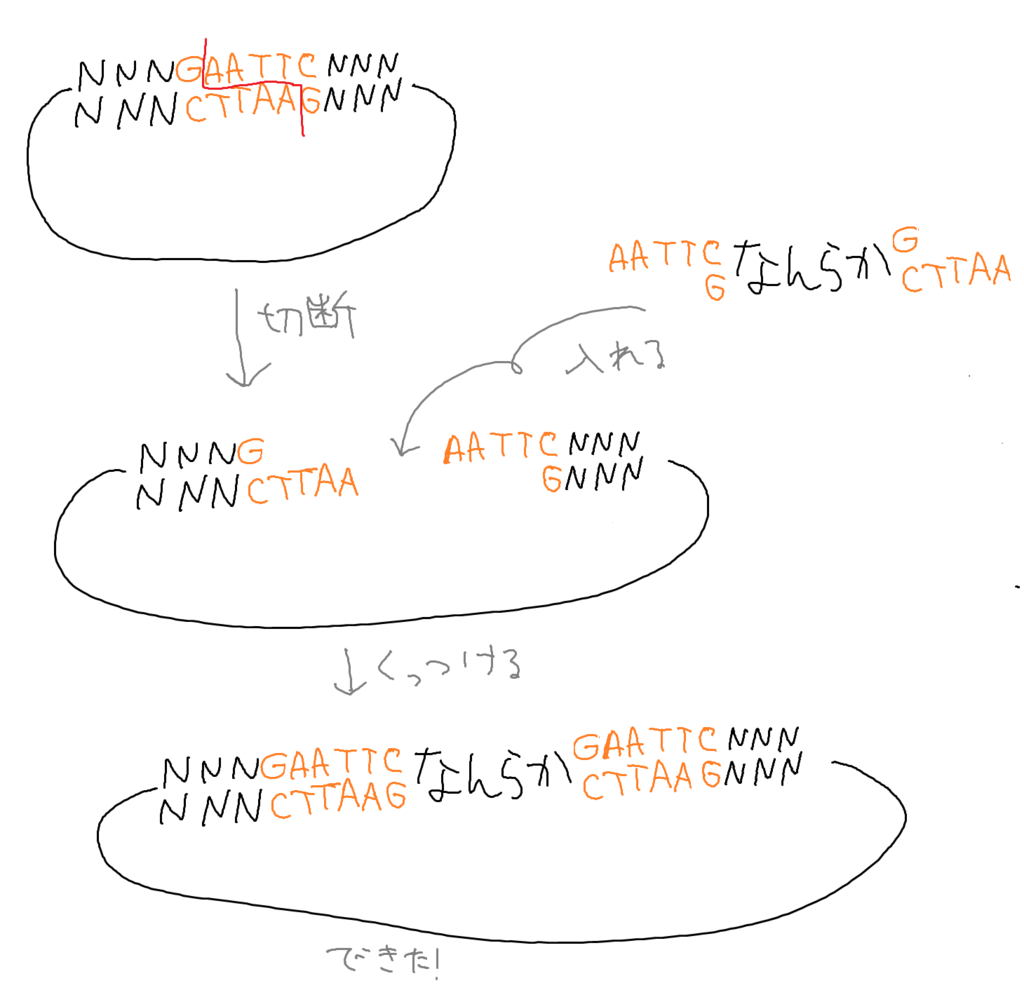
上の図が概略です。詳しい説明をしていきます。
- ある制限酵素認識サイトを一つだけ持ったプラスミドを用意します。(ここでは例としてEcoR1認識サイトで考えています。塩基配列のNはATGCのうちどれでもいいということを表す記号です。)
- その制限酵素認識サイトを制限酵素で切断します。
- 同じ制限酵素で両端を切断したなんらかの遺伝子を含む直鎖上DNA(環状でないDNA)を用意します。このDNAとプラスミドを切ったものは、それぞれの末端の一本鎖の部分が相補的になっているために、うまいこと環状になるように結合することができます。(うまいこと挿入された環状DNA以外にもいろいろなパターンのものができます)
- それぞれの鎖の切断されている部分を結合させる酵素を用いて完全なプラスミドを作り完成です。
これだけ聞くとなんかこの方法でうまいこと行きそうですが、実際のところこのシステムではいろいろ不都合なことがあります。例として、ある遺伝子ひとつが入ったプラスミドを作った後、同じ方法を使ってそのプラスミドにさらに別の遺伝子ひとつを入れることがうまくできません。
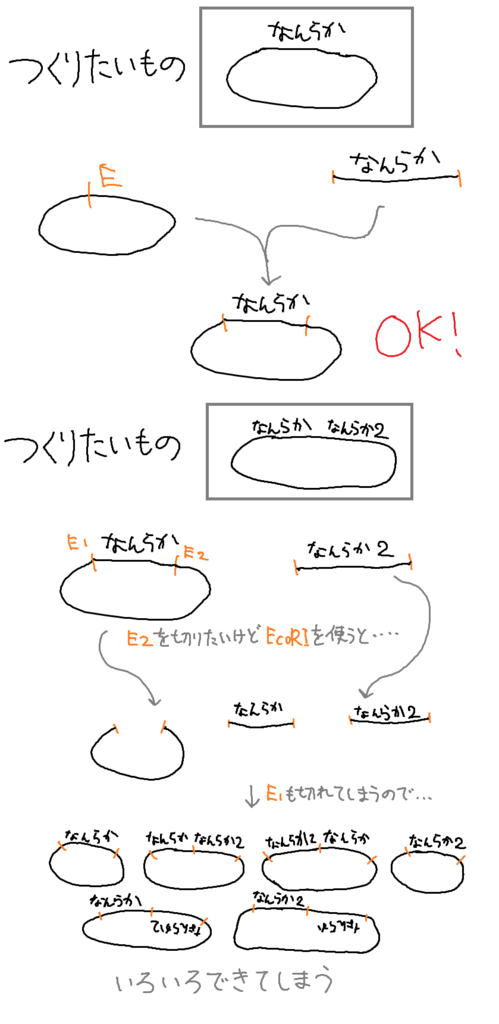
うまくいきません
図のように、一つの制限酵素サイトを切断してそこに遺伝子を挿入する方法では、作りたいもの以外にもいろいろなものができてしまします(図中に示した以外のものもいろいろできます)。このようにいろいろできてしまったものを選別するのは結構大変です。ではどうすればこの問題を解決できるでしょうか。
XとSの特性
先ほどの問題は複数の制限酵素認識サイトをうまく使うと解決できます。まずは次の図を見てください。
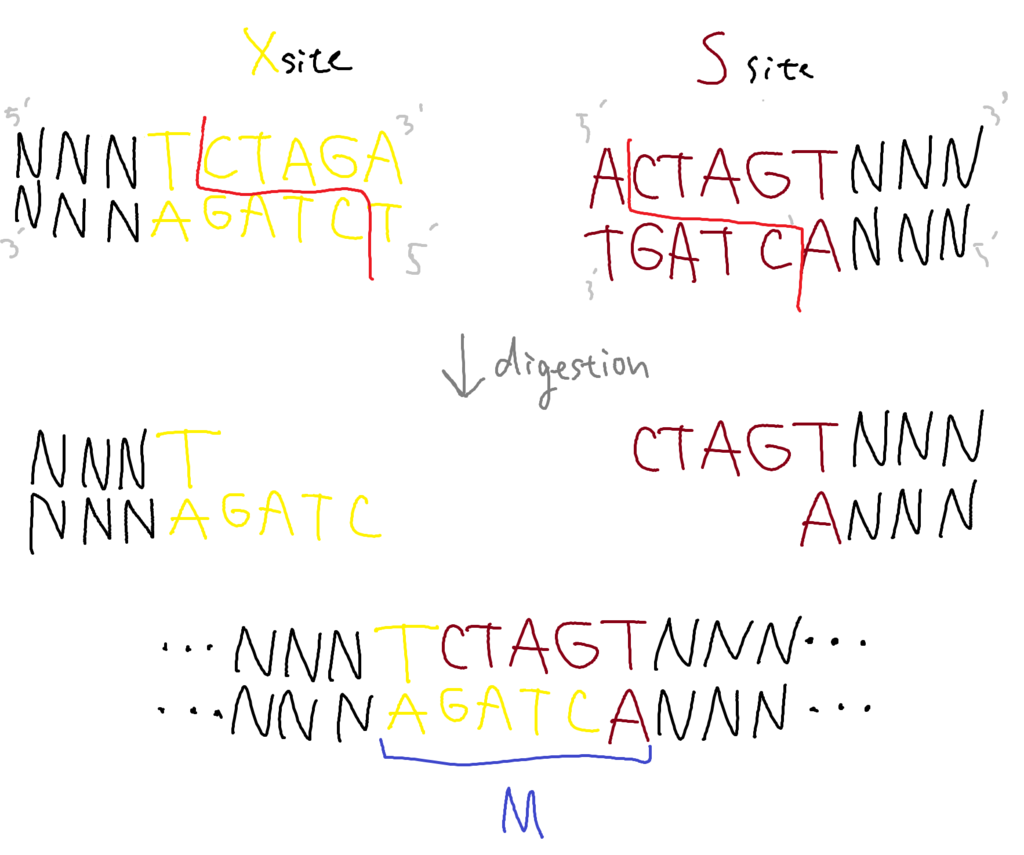
Xba1認識サイトとSpe1認識サイトを眺めてみるとわかることですが、それぞれを対応する制限酵素で切断した時に末端にできる一本鎖部分は共通しています。つまり、Xba1で切ったものととSpe1で切ったものを結合させることができるというわけです。さらに、そうして結合させてできた部分M(たぶんmixの略)はもうどちらの酵素にも認識されません。この特性を生かすと、プラスミドへの遺伝子の追加をうまいことやることができます。
BioBrick Standard Assembly
では具体的な方法を見ていきます。ここで紹介するのがBiobrick規格のプラスミドを使って遺伝子をくっつけていくときに実際に使われる方法(の一つ)です。ちょっと前に紹介したように、BioBrickの規格にそったプラスミドでは、目的の遺伝子はEXサイトとSPサイトの間に存在しています。
下の図は、「い」と「ろ」という遺伝子がそれぞれ目的遺伝子として入っているBioBrickプラスミドを使って「いろ」が入ったプラスミドを作る方法を解説したものです。
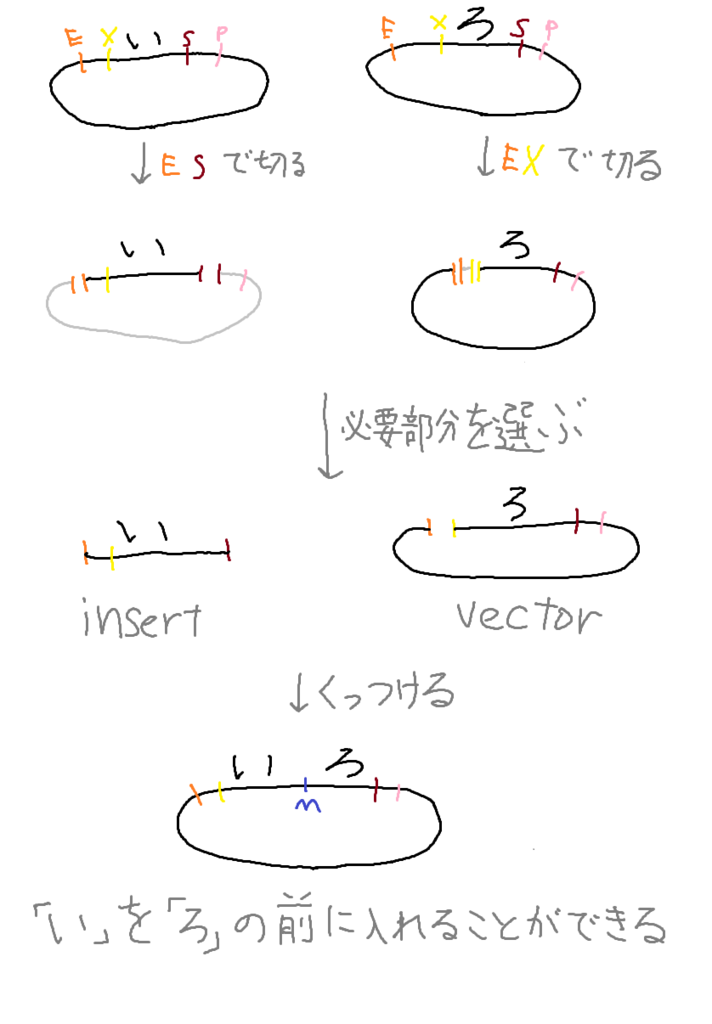
ここでは、「い」の遺伝子を「ろ」の遺伝子が入ったプラスミドに入れています(「ろ」の遺伝子を「い」の遺伝子が入ったプラスミドに入れることも可能です)。
- 「い」プラスミドをEcoR1とSpe1で、「ろ」プラスミドをEcoR1とXba1で、それぞれ制限酵素処理します。
- それぞれ断片が2つずつできますが、目的遺伝子を含んだほうの断片をそれぞれ使います。目的遺伝子を含んだ断片がプラスミドの大部分を含んでいない場合それをinsertと呼び、プラスミドの大部分を含んでいる場合それをvectorと呼びます。
- insertとvectorを結合させます。EサイトはEサイト同士で結合し、前述のとおりXサイトはSサイトと結合します。
- 結果として「いろ」が入ったプラスミドを作ることができます。Mサイトは4つの制限酵素に認識されないために、このプラスミドもまたBioBrick規格に沿ったものになっています。
この最終産物としてできた目的のプラスミドもまたBioBrick規格を満たしているというのが重要です。BioBrick規格を満たしているプラスミド間では同じような操作ができるために、この方法を使えばどんどん遺伝子を付け足していくことができます。その確認の意味も込めて、今度は「は」という遺伝子を、今作った「いろ」プラスミドの「いろ」遺伝子の後ろに挿入する方法を見てみましょう。
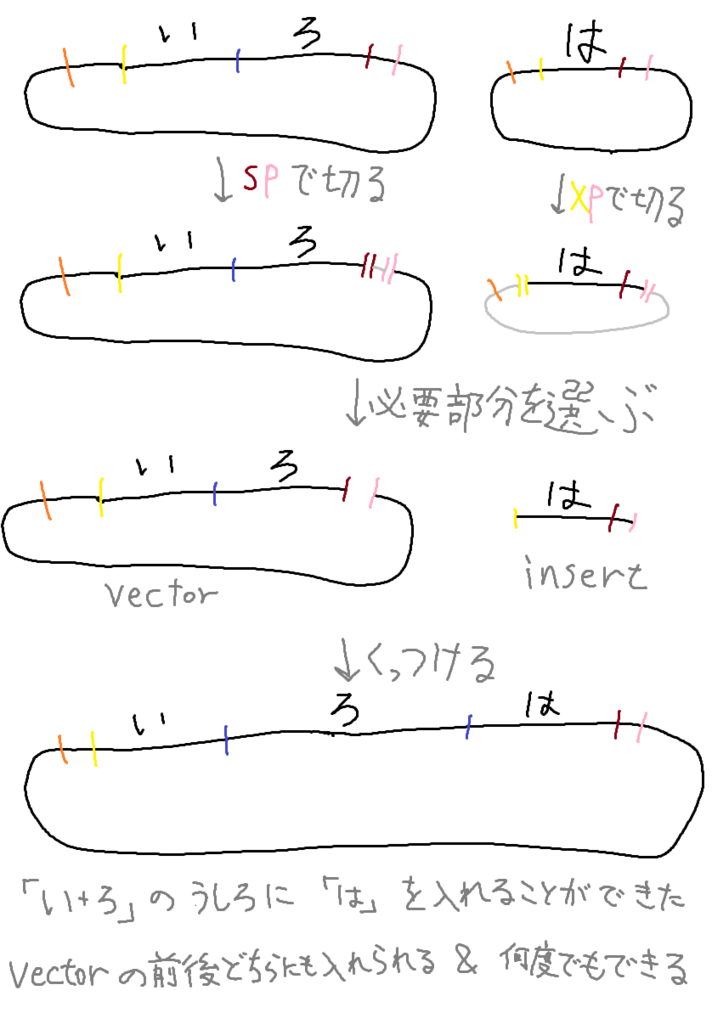
insertをvectorの遺伝子の後ろに挿入するためにはXba1, Spe1, Pst1の3つを使います。先ほど使わなかったPst1はこの状況のためにあったわけです。Mサイトが4つの制限酵素で認識されないため、それに全く気を遣わずに操作を行うことができることがわかると思います。
以上でBioBrick規格とそれに付随する実験操作や概念の説明は終わりです。うまいことできていますね。BioBrick規格のプラスミドはいろんな種類のものがあればあるほどその効果を発揮するため、iGEMではBioBrick規格の新たなプラスミドを作って団体が管理するRegistryに追加することが大きな目標になっています。
BioBrickの規格はこれで全てではありませんが、理解が必要で面白い部分は大体この記事で説明したので全部です(個人の感想です)。次回からはこの規格を生かしてどのように実験を進めていくか説明をしていきたいと思います。